Le projet PPG-3
Paléoparasitologie et paléogénétique, à la recherche des cryptosporidies anciennes
Le projet PPG-3 (Paléoparasitologie et Paléogénétique – Phase 3) associe des chercheurs de l’UMR Chrono-environnement et de l’UMR PAM (INRAE, Dijon), et porte sur la détection de l’ADN ancien (ADNa) de parasites unicellulaires du genre Cryptosporidium, qui infestent le système digestif d’une grande variété d’hôtes animaux, dont l’espèce humaine. La détection de ce parasite en contexte paléoenvironnemental ou archéologique permet d’aborder de multiples problématiques comme le rôle des activités humaines passées (domestication, anthropisation du territoire, gestion des déchets, migrations…) dans l’émergence et la distribution d’espèces pathogènes à notre époque.
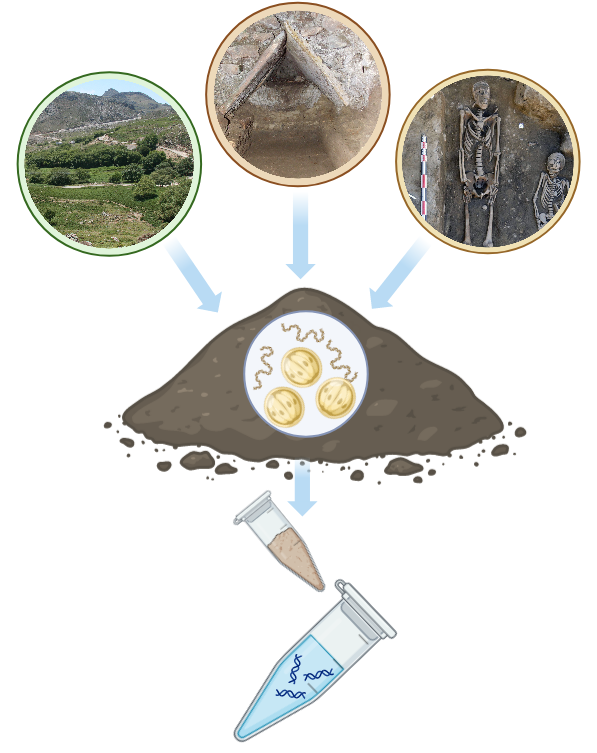
Les restes observables de parasites peuvent être de différentes sortes en fonction de leur physiologie et de leur écologie, et provenir d’une variété de contextes chrono-culturels et d’échantillonnage. Afin de détecter ces traces dans les environnements et sociétés anciennes au cours du temps, le projet PPG-3 s’intéresse tout particulièrement aux matrices sédimentaires qui peuvent avoir conservé les restes microscopiques et moléculaires des cryptosporidies y ayant autrefois été disséminées, ainsi qu’aux coprolithes ou paléofèces. Ces sédiments/coprolithes peuvent provenir du carottage opéré dans un bassin versant fréquenté par des animaux d’élevage pendant de longues périodes historiques, de structures archéologiques comme des égouts ou des latrines anciennes, ou bien encore de sépultures qui autorisent une approche paléo-épidémiologique. Ces échantillons ont toutes les chances d’avoir conservé les traces de la matière fécale dont ils étaient autrefois tout ou partie constitués, et a fortiori, celles de parasites gastro-intestinaux, dont les kystes microscopiques du Cryptosporidium et des fragments de son génome.
Une revue de la littérature récente (Roche et al., 2023) dresse un état des lieux des détections de ce parasite dans le registre paléo/archéologique. On peut y constater qu’étonnement, très peu de travaux ont été publiés concernant ce parasite en particulier et qu’ils n’ont que très peu porté sur la détection d’ADNa mais plutôt sur celle de paléoantigènes à l’aide de kits médicaux d’immunodiagnostic qui n’ont pas été optimisés pour l’analyse d’échantillons anciens.
Le développement d’outils spécialement adaptés à la recherche de molécules d’ADN très dégradées (« taphonomisées ») et à une plus grande diversité d’espèces (au-delà des taxons visés par les kits médicaux) semble donc une première étape essentielle à une meilleure compréhension de l’émergence et de la diffusion des cryptosporidies au cours du temps et aux interactions qui les ont permises.