RAMA
Projet RAMA
Émergence de la résistance bactérienne aux antibiotiques dans des microbiotes digestifs de patients traités par anticancéreux (RAMA)
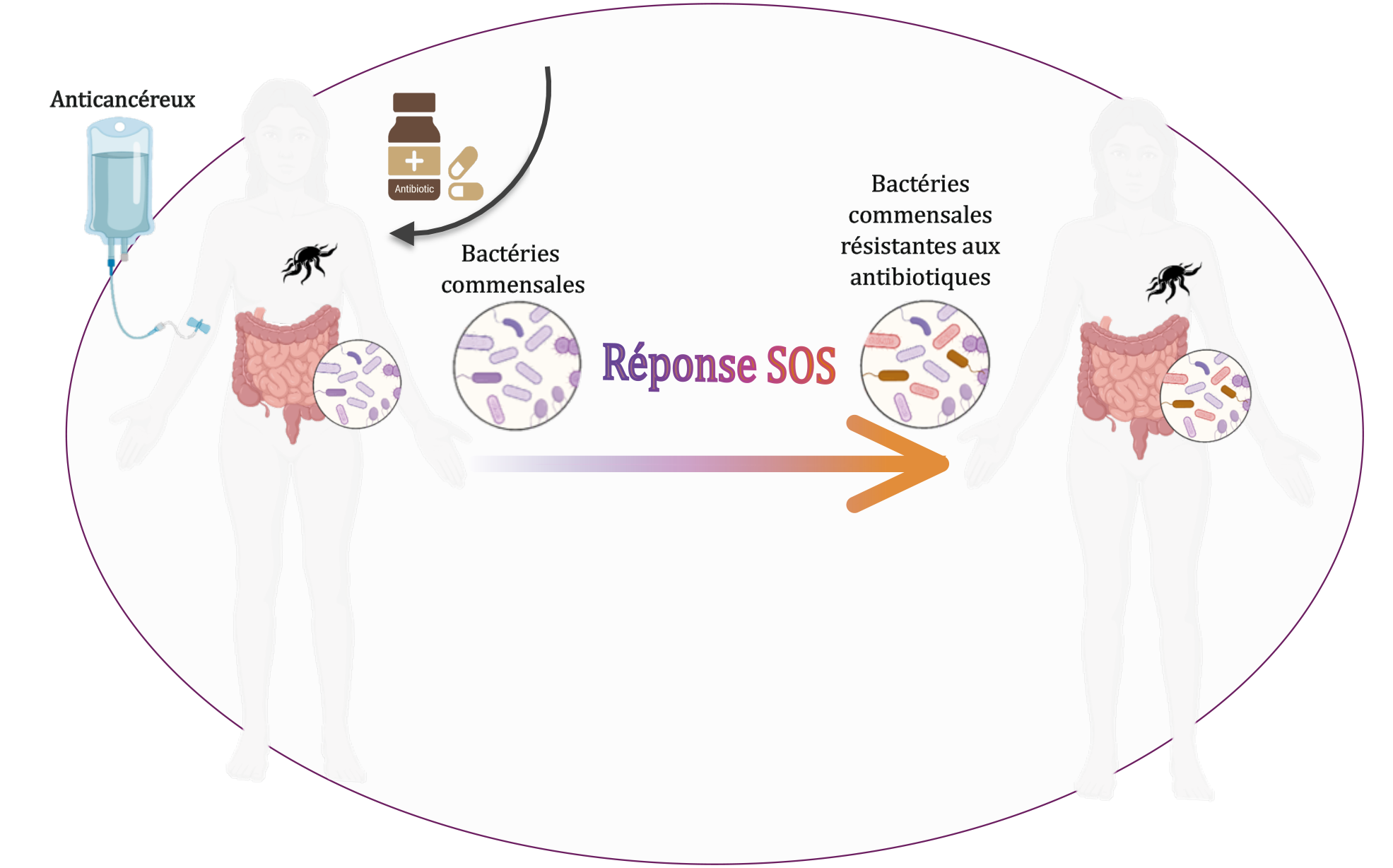
Les cancers solides sont traités avec médicaments qui attaquent l'ADN des cellules cancéreuses et des bactéries du microbiote digestif. Avec le projet RAMA, nous souhaitons tester si les médicaments anticancéreux favorisent le développement de bactéries résistantes aux antibiotiques en utilisant différents modèles : chez des patients traités par des cytotoxiques, dans un modèle in vitro de microbiote digestif et un modèle murin de microbiote humanisé.
Le traitement des cancers solides consiste fréquemment en l’utilisation de médicaments cytotoxiques, lesquels ciblent l’ADN des cellules cancéreuses et des bactéries du microbiote digestif. Notre équipe a déjà montré in vitro que des médicaments anticancéreux connus pour activer la réponse SOS bactérienne, quand ils sont donnés en doses thérapeutiques, augmentent le taux de mutation bactérienne et l’émergence de bactéries résistantes aux antibiotiques.
Dans ce contexte, nous mesurerons l’influence du traitement par des molécules anticancéreuses connues pour activer la réponse SOS bactérienne sur l’émergence de bactéries commensales résistantes aux antibiotiques dans le microbiote digestif de patients cancéreux. Les résultats obtenus en clinique seront investigués dans deux modèles expérimentaux : un modèle in vitro de microbiote digestif et un modèle murin de microbiote humanisé.
Nous mesurerons l’impact des traitements anticancéreux sur l’apparition de bactéries commensales résistantes aux antibiotiques. Ainsi, nous quantifierons la fréquence d’émergence des mutants de bactéries commensales du tube digestif résistants aux antibiotiques à large spectre utilisés lors du traitement. Nous testerons (1) quatre molécules cytotoxiques – deux molécules inductrices de la réponse SOS bactérienne et deux molécules contrôles non inductrices, (2) en combinaison avec la ceftriaxone, une céphalosporine de 3e génération. Cette séquence de traitement est souvent prescrite aux patients atteints de cancers. Les cytotoxiques testés ont été choisis en raison de l’importance de la file active de patients traités par ces quatre molécules. Nous quantifierons ensuite l’émergence de bactéries commensales résistantes aux antibiotiques (par culture sur géloses sélectives) et mesurerons les modifications du microbiote par métagénomique 16S (exploration des variations de compositions) et par métagénomique shotgun (exploration des variations de fonctions bactériennes).
Dans ce contexte, nous mesurerons l’influence du traitement par des molécules anticancéreuses connues pour activer la réponse SOS bactérienne sur l’émergence de bactéries commensales résistantes aux antibiotiques dans le microbiote digestif de patients cancéreux. Les résultats obtenus en clinique seront investigués dans deux modèles expérimentaux : un modèle in vitro de microbiote digestif et un modèle murin de microbiote humanisé.
Nous mesurerons l’impact des traitements anticancéreux sur l’apparition de bactéries commensales résistantes aux antibiotiques. Ainsi, nous quantifierons la fréquence d’émergence des mutants de bactéries commensales du tube digestif résistants aux antibiotiques à large spectre utilisés lors du traitement. Nous testerons (1) quatre molécules cytotoxiques – deux molécules inductrices de la réponse SOS bactérienne et deux molécules contrôles non inductrices, (2) en combinaison avec la ceftriaxone, une céphalosporine de 3e génération. Cette séquence de traitement est souvent prescrite aux patients atteints de cancers. Les cytotoxiques testés ont été choisis en raison de l’importance de la file active de patients traités par ces quatre molécules. Nous quantifierons ensuite l’émergence de bactéries commensales résistantes aux antibiotiques (par culture sur géloses sélectives) et mesurerons les modifications du microbiote par métagénomique 16S (exploration des variations de compositions) et par métagénomique shotgun (exploration des variations de fonctions bactériennes).
En bref
Durée du projet : du 01/01/2025 au 01/07/2027
Financement : Ligue contre le Cancer – Bourgogne Franche-Comté
Coordination du projet et personne référente au sein du laboratoire :
- Didier Hocquet, Professeur – UMLP, PATHOGENESdhocquet@-Code to remove to avoid SPAM-univ-fcomte.fr, +33 (0)3 70 63 21 34, bureau R212 (HdC)
Autres membres du laboratoire impliqués :
Personnels recrutés :
- Laura Camila CARRERA PAÉZ (postdoc) : Gestion et analyse des prélèvements cliniques, modèle in vitro, modèle murin.
Partenaires et laboratoires associés :
- Pr Sylvain LADOIRE et Anne-Laure REROLE, CGFL – Dijon
- Dr Dewi VERNEREY, UMQVC – CHU de Besançon
- Dr Marie KROEMER, Pharmacie Centrale – CHU de Besançon
- Dr Maïder PAGADOY, Centre d’Investigation Clinique – CHU de Besançon
En savoir plus
Désolé, pas d'actualité pour l'instant.
Références bibliographiques :
- Alexandre Meunier, Virginie Nerich, Christine Fagnoni-Legat, Marion Richard, Didier Mazel, et al.. Enhanced emergence of antibiotic-resistant pathogenic bacteria after in vitro induction with cancer chemotherapy drugs. Journal of Antimicrobial Chemotherapy, 2019, 74 (6), pp.1572-1577. ⟨10.1093/jac/dkz070⟩. ⟨hal-02083466⟩
Plateformes technologiques associées
Big Data et Bioinformatique au Service de la Santé (2B2S)
UFR Santé et CHU de Besançon
Crédits image du bandeau : légendes