PARADE
Projet PARADE
Pseudomonas Anti-phage Resilience and Defence among Epidemic clones
Les phages sont des virus qui tuent spécifiquement les bactéries. Ils jouent un rôle crucial dans le contrôle des populations bactériennes dans les écosystèmes. Par un phénomène de coévolution, les bactéries ont développé une grande diversité de défenses anti-phages. Nous émettons l'hypothèse que le succès de certains clones épidémiques qui sont répandus à l'échelle mondiale doivent leur succès, au moins en partie, à leur plus grande résistance aux phages.
Les phages infectent les bactéries en injectant leur matériel génétique ce qui provoque la lyse cellulaire. Très répandus dans les environnements telluriques, aqueux et dans les égouts, ils sont spécifiques d’une espèce bactérienne, voire d’une population clonale.
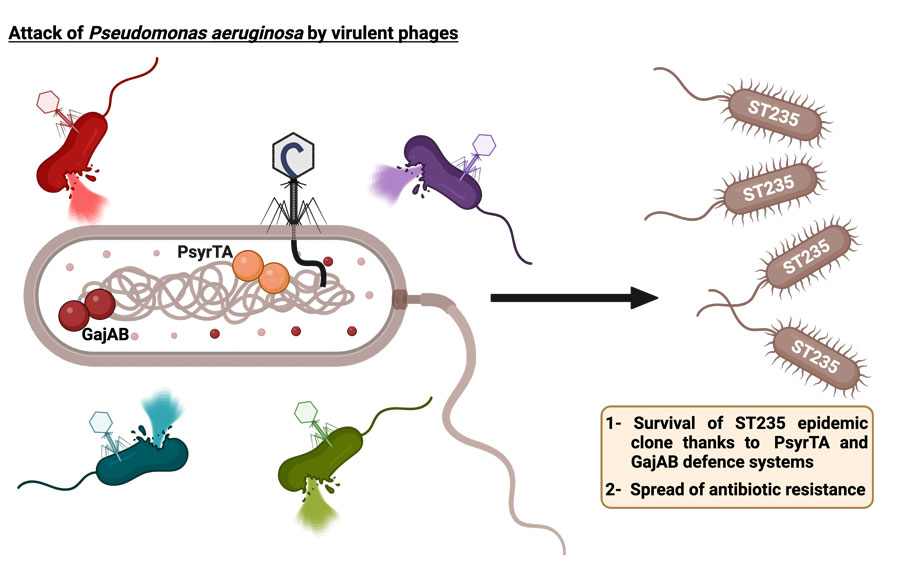
Le clone ST235 de P. aeruginosa, dont la diffusion est mondiale, fait partie des clones à haut risque épidémique. Particulièrement virulent en raison de la production de la toxine ExoU, ce clone est associé à une faible chance de succès thérapeutique en raison de son profil hautement résistant aux antibiotiques, résultat de l’acquisition de gènes par transfert horizontal.
Des travaux préliminaires menés au laboratoire Chrono-environnement ont montré que ce clone pourrait devoir sa capacité accrue de survie dans l’environnement en échappant à la prédation des bactériophages (données non publiées) car (1) il est beaucoup plus résistant aux phages que la souche de référence P. aeruginosa PAO1, (2) il présente un cluster de 10 gènes (cluster 2) prédit pour coder des systèmes de défense anti-phages en particulier les systèmes Gabija (clivage de l’ADN exogène) et PsyrT/PsyrA (système suicide toxine/antitoxine), et (3) l’inactivation de ce cluster 2 rend ce clone sensible aux phages.
Persistance du clone épidémique ST235 en raison de ses systèmes anti-phages GajAB et PsyrTA. Image créée avec Biorender.
Dans ce contexte, le but de ce projet est :
- d’étudier l’impact sur la défense anti-phage des systèmes Gabija (clivage de l’ADN exogène) et PsyrT/PsyrA (toxine/antitoxine) présents chez le clone épidémique H27244 (ST235),
- d’étudier la répartition de ces systèmes dans d’autres ST de P. aeruginosa épidémiques et non-épidémiques,
- de mesurer le niveau d’expression de ces systèmes chez différents ST,
- d’étudier la nature des phages ciblés par ces systèmes de défense,
- d’étudier la compatibilité des systèmes entre eux et déterminer s’ils peuvent avoir un effet synergique.
En bref
Durée du projet : du 01/09/2025 au 01/09/2027
Financement : HARMI
Personnes référentes au sein du laboratoire :
- Didier Hocquet, Professeur – UMLP, PATHOGENESdhocquet@-Code to remove to avoid SPAM-univ-fcomte.fr, +33 (0)3 70 63 21 34, bureau R212 (HdC)
- Catherine Llanes, Professeure – UMLP, PATHOGENES, Service communicationchrono-env.direction@-Code to remove to avoid SPAM-univ-fcomte.fr, +33 (0)3 63 08 22 76, bureau R222 (HdC)
Coordination du projet : Catherine Llanes
Autres membres du laboratoire impliqués :
Personnels recrutés :
- Olympe Bonnardel (thèse)
Partenaires et laboratoires associés :
- Gregory Resch (Centre Hospitalier Universitaire Vaudois [CHUV], Centre de Recherche et d’Innovation en Sciences Pharmaceutiques Cliniques [CRISP], Laboratoire des bactériophages et de phagothérapie, Lausanne, Suisse